15日ごと変異、陰謀論否定 遺伝子技術で見るコロナ
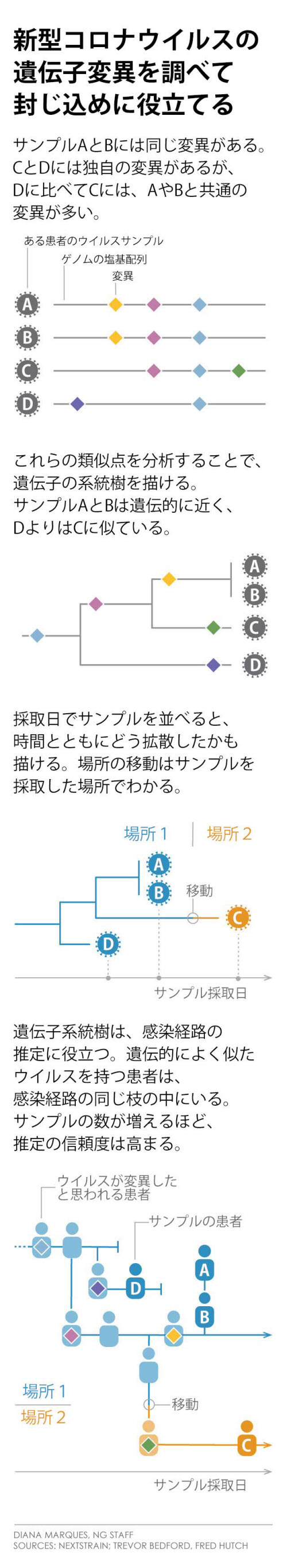
無償で公開されているオープンソースプロジェクト「ネクストストレイン」(Nextstrain.org)は、アウトブレイク(集団感染)を起こした病原体の博物館のようなものだ。世界各地の研究機関が、患者から採取したウイルスの遺伝子配列データをここに投稿する。ネクストストレインはそのデータを使って、感染の広がり方を示した世界地図や、ウイルスの系統樹を描き出している。
ネクストストレインが取りこんだ新型コロナウイルスのゲノム(全遺伝情報)は、2020年3月末の時点で2000を超えた。データからは、感染の拡大とともにウイルスが平均15日ごとに変異していることが明らかになっている。
「変異」などと聞くと恐ろしげな想像をしてしまうかもしれないが、ウイルスの有害さが増しているという意味ではない。単に変化しているというだけで、むしろウイルスがどこから来たのかを迅速に知ることができることにくわえて、起源に関する妄言も否定できる。
「これらの変異は完全に無害で、ウイルスの拡散の仕方を解明するパズルのピースとして役に立ちます」。ネクストストレインの共同設立者で、米ワシントン州シアトルにあるフレッド・ハッチンソンがん研究センターの計算生物学者トレバー・ベッドフォード氏はそう話す。
新型コロナウイルスを追跡するこの手法は、深刻な世界的な流行(パンデミック)の報道があふれる中で、ひときわ明るく輝いている。同様の手法は、過去にジカ熱やエボラ熱などの感染症が流行した際にも、重要な役割を担っていた。
しかも、遺伝子解析のコストの低下とスピードや効率の向上により、世界各地の研究者がごく少人数で、新型コロナウイルスの感染経路をこれまで以上に速やかに解明できるようになった。その結果は、特に検査が追いつかない場所で、感染を封じ込める戦略から感染ペースを緩和する戦略に移行するべきか否かを判断するのに役立つはずだ。
「5年前にエボラ熱が流行したときには、サンプルを採取してから、ゲノムの塩基配列を決定しデータを公開するまでに1年かかりました」とベッドフォード氏は言う。「今では2日から1週間でできるようになりました。これらの技術をリアルタイムに利用してアウトブレイク対策に生かせるようになったのは、今回が初めてです」
「知らないうちに広めうる」ことを証明
ベッドフォード氏の研究室が新型コロナウイルスを追跡し始めたのは、2月から3月にかけてワシントン州で感染者が増えだした頃だった。公衆衛生当局は当時、患者の旅行歴を追跡し、接触した疑いのある人を突き止めることに注力していた。
一方、ベッドフォード氏らは、20人以上の患者の鼻から採取したサンプルを分析し、遺伝子の解読を始めた。新型コロナウイルスがどこで、どのように変化したかを追跡していくと、驚くべき事実が明らかになった。シアトルで最初に感染者が見つかった1月21日以降、何週間もの間、シアトルの人々の間でウイルスがいわばひっそりと培養され続けていたのだ。最初の患者は、中国の武漢を訪れた35歳の人物だった。
別の言い方をすると、軽症で病院にかからなかった人や、検査を受けていなかった人々が、知らないうちに新型コロナウイルスを広めうることが科学的に証明された。この発見から、感染スピードを遅らせる都市封鎖、学校・職場・店舗などの閉鎖、他人と一定の「社会的距離」を保つなどの対策が、世界中で次々と打ち出されることになった。
「ゲノムデータは、ウイルス感染がどのように拡大するかについて、より多くのことを教えてくれます」とベッドフォード氏は話す。
ネクストストレインの可視化ツールは、新型コロナウイルスについて知りたいという市民の欲求も満たしている。そう評価するのは、米カリフォルニア州にあるスクリプス研究所の計算生物学者で、西ナイルウイルスやジカウイルスなど1000種類以上のゲノムデータをネクストストレインに投稿しているクリスチャン・アンダーセン氏だ。
「これまでウイルスの遺伝子系統樹なんて眺めているのは私のようなオタクだけだったのに、今ではツイッター上にあふれかえっています。私がネクストストレインのようなツールを気に入っているのは、こういうところです」と氏は話す。
このサイトのオープンな姿勢も、ゲノムデータをぜひ共有したいと世界中の研究者に思わせる理由の1つになっている。アンダーセン氏の研究室にウイルスのサンプルを送りたいと言ってくる研究者や、解析の方法について助言を求めてくる研究者もいる。「画面に表示されるデータを見て、『うちにも患者がいるから遺伝子解析したい』と言うのです」
データを可視化した図や系統樹は、パンデミックの広がりの全体像を見るには便利だ。だがアンダーセン氏は、たまたまこれらを目にした人々が、背景にある広範なデータを検討せずに何らかの結論に飛びつこうとすることに警鐘を鳴らしている。
実際、ベッドフォード氏もその1人だった。イタリアで確認されたドイツ人感染者のサンプルと、その1カ月前に感染したミュンヘンの患者のサンプルで遺伝子配列データがよく似ていることから、ヨーロッパでのアウトブレイクはドイツから始まったことが示唆される、とする内容をツイッターに投稿したものの、氏はのちに撤回した。
「系統樹はつながりを示唆するものではありますが、欠けているピースが非常に多く、何が起きたのかを説明する方法は複数ありえます」とアンダーセン氏は警告する。
ベッドフォード氏は、検査や症例の監視が十分に行われていない場所では、社会的距離を保持する戦略が有効に機能しているかどうかを検証するうえで、今後も遺伝子データが手がかりになると指摘する。
「感染がどのくらい減少するかがわかり、『規制を緩めてもよいか?』という問いへの答えが得られます」と氏は言う。
2019年11月頃に動物からヒトへ飛び移った
ウイルスの進化の歴史を明らかにするネクストストレインの能力はまた、「新型コロナウイルスは生物兵器として実験室で秘密裏に製造されたものである」という陰謀論を即座に打ち砕いた。
3月17日付で学術誌「ネイチャー・メディシン」に発表されたアンダーセン氏らの論文は、新型コロナウイルスのゲノムの特徴を、重症急性呼吸器症候群(SARS)や中東呼吸器症候群(MERS)を含むコロナウイルスや、コウモリやセンザンコウなどの動物から単離されたコロナウイルス株などのゲノムの特徴と比較して、陰謀論を否定した。
そもそも、新型コロナウイルスの基本的な構造は、これまでに研究されてきたどのコロナウイルスとも異なっている。また、その遺伝子には、実験室で培養されたのではく、生きた免疫系と戦ってきたことを示唆する特徴がある。
それだけではない。生物兵器の設計者は、最大の効果を得るために、既存のウイルスをヒントにして完璧なウイルスを作り出そうとするだろう。ところが新型コロナウイルスには、自然選択によるものと思われる小さな欠陥がいくつかある。
例えば、細胞への感染の仕方だ。コロナウイルスが動物細胞に感染するときには、ブロッコリーの頭の部分のような形の「スパイク」と呼ばれるタンパク質を使って細胞の「出入口」にあたる受容体に結合し、細胞内に侵入する。新型コロナウイルスは、ヒト細胞の「ACE2(アンジオテンシン変換酵素2)」を利用して細胞に侵入するが、この作用が最適なものではないことが実験によって示されている。
「人為的に完璧なウイルスを作ろうとするなら、このようにはしないでしょう」とアンダーセン氏は言う。氏らの分析結果は、新型コロナウイルスが2019年11月頃に動物からヒトへ飛び移ったことを示唆している。
将来的には、新しいウイルスが出現した際に、感染が発生した場所や地域を拡散前に特定する上で、遺伝子解析技術がさらに重要な役割を果たすようになるだろう。
「例えば、アフリカのとあるコミュニティーで新しいウイルスが出現したとしても、今ならそのサンプルを実験室に運んで、ショットガン法という遺伝子解析を行えます」とフィル・フェッボ氏は話す。この方法は、ランダムな短い断片の塩基配列を読み取ってゲノム全体を決定できるため、当局は、感染拡大を阻止する戦略を迅速に定められる。なお氏は内科医であり、米カリフォルニア州サンディエゴに拠点を置く遺伝子解析機器の世界最大手イルミナ社の最高医療責任者でもある。
迅速に対応できるウイルス監視網を世界中に張り巡らせるためには、まだたくさんの仕事をしなければならない。各国政府を巻き込み、実験室を設置し、解析装置を操作して結果を解釈できるスタッフを募集し、訓練する必要がある。
「問題は、技術的制約ではありません。国際コミュニティーの覚悟なのです」とフェッボ氏は言う。
(文 SARAH ELIZABETH RICHARDS、訳 三枝小夜子、日経ナショナル ジオグラフィック社)
[ナショナル ジオグラフィック ニュース 2020年3月31日付]
ワークスタイルや暮らし・家計管理に役立つノウハウなどをまとめています。
※ NIKKEI STYLE は2023年にリニューアルしました。これまでに公開したコンテンツのほとんどは日経電子版などで引き続きご覧いただけます。